Esta web utiliza cookies para que podamos ofrecerte la mejor experiencia de usuario posible. La información de las cookies se almacena en tu navegador y realiza funciones tales como reconocerte cuando vuelves a nuestra web o ayudar a nuestro equipo a comprender qué secciones de la web encuentras más interesantes y útiles.
Los dominios proteicos BuD podrían ser utilizados como ‘drones’ para dirigir proteínas reparadoras o activadoras del ADN hacia secuencias específicas del genoma
Esta nueva tecnología podría servir para hacer modificaciones ‘a la carta’ del genoma, destinadas a reparar daños genéticos o a transformar microorganismos para la generación de biocombustibles
El trabajo, que ha despertado el interés de varias compañías internacionales, lo presenta el último número de la revista 'Acta Crystallographica Section D: Biological Crystallography'
El Grupo de Cristalografía de Macromoléculas del Centro Nacional de Investigaciones Oncológicas (CNIO) ha conseguido reprogramar la unión al ADN de una proteína denominada BuD para reconducirla hacia regiones específicas del genoma. Para Guillermo Montoya, el investigador que ha liderado el estudio, este descubrimiento “permitirá hacer modificaciones y editar las instrucciones contenidas en el genoma para el tratamiento de enfermedades genéticas o el desarrollo de organismos modificados genéticamente”. El trabajo lo publica el último número de la revista Acta Crystallographica Section D: Biological Crystallography.
La posibilidad de hacer modificaciones a la carta en los genes de los organismos vivos tiene aplicaciones muy diversas, que abarcan desde el tratamiento de enfermedades humanas hasta la biología sintética, disciplina que busca la creación o mejora de los organismos vivos ya existentes para su uso biotecnológico
Para conseguirlo, varios investigadores de todo el mundo han puesto el foco de atención en las proteínas que se unen de forma específica al ADN: una vez reprogramadas para dirigirlas hacia puntos concretos del genoma, podrían unirse a efectores del material genético (proteínas reparadoras o activadoras del ADN, entre otras) para poder modificar sus instrucciones o rediseñar los circuitos genéticos a voluntad.
En esta tarea, el equipo del CNIO ha descifrado el código de unión al ADN de BurrH, una nueva proteína identificada en la bacteria Burholderia rhizoxinica cuyo dominio BuD se une específicamente al genoma. Para ello, los investigadores han resuelto la estructura tridimensional de la proteína completa mediante la técnica biofísica de cristalografía de rayos- X.
La principal ventaja de los BuDs proviene de su alta especificidad: son capaces de distinguir secuencias de ADN que difieren únicamente en dos nucleótidos (letras que componen el ADN). “Esta alta especificidad actúa como un GPS que les permite encontrar sus destinos dentro del intrincado mapa del genoma”, explica Montoya, y añade: “Son muy versátiles y fáciles de reprogramar en comparación con otras proteínas utilizadas para este mismo objetivo”.
El Grupo de Montoya ha rediseñado BuDs capaces de reconocer las regiones del genoma cercanas a las mutaciones responsables de la anemia falciforme, una patología causada por modificaciones en el gen de la globina beta y que produce alteraciones en los glóbulos rojos de la sangre. “La unión de proteínas reparadoras del ADN a estos BuDs rediseñados podría servir para corregir las alteraciones genéticas de los pacientes con esta enfermedad”, afirman los investigadores.
Montoya asegura que son varias las empresas que están ya interesadas en esta nueva tecnología. “Nuestra herramienta podría servir, además de para el tratamiento de enfermedades genéticas, para la modificación genética de microorganismos destinada a la síntesis de metabolitos necesarios para [por ejemplo] la producción de biocombustibles”, asegura.
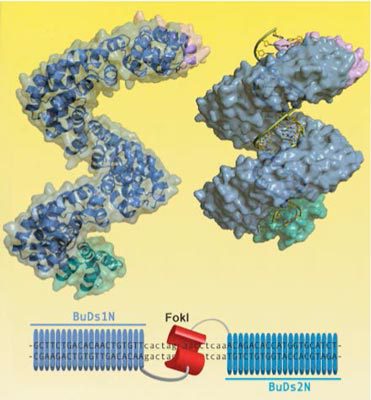
Cambio de estructura de los BuDs, antes (izquierda) y después (derecha) de reconocer una secuencia específica de ADN. /Guillermo Montoya. CNIO
Artículo de referencia
BuD, a helix-loop-helix DNA-binding domain for genome modification. S. Stella, R. Molina, B. López-Méndez, A. Juillerat, C. Bertonati, F. Daboussi, R. Campos-Olivas, P. Duchateau, G. Montoya. Acta Cryst. (2014). doi: 10.1107/S1399004714011183
